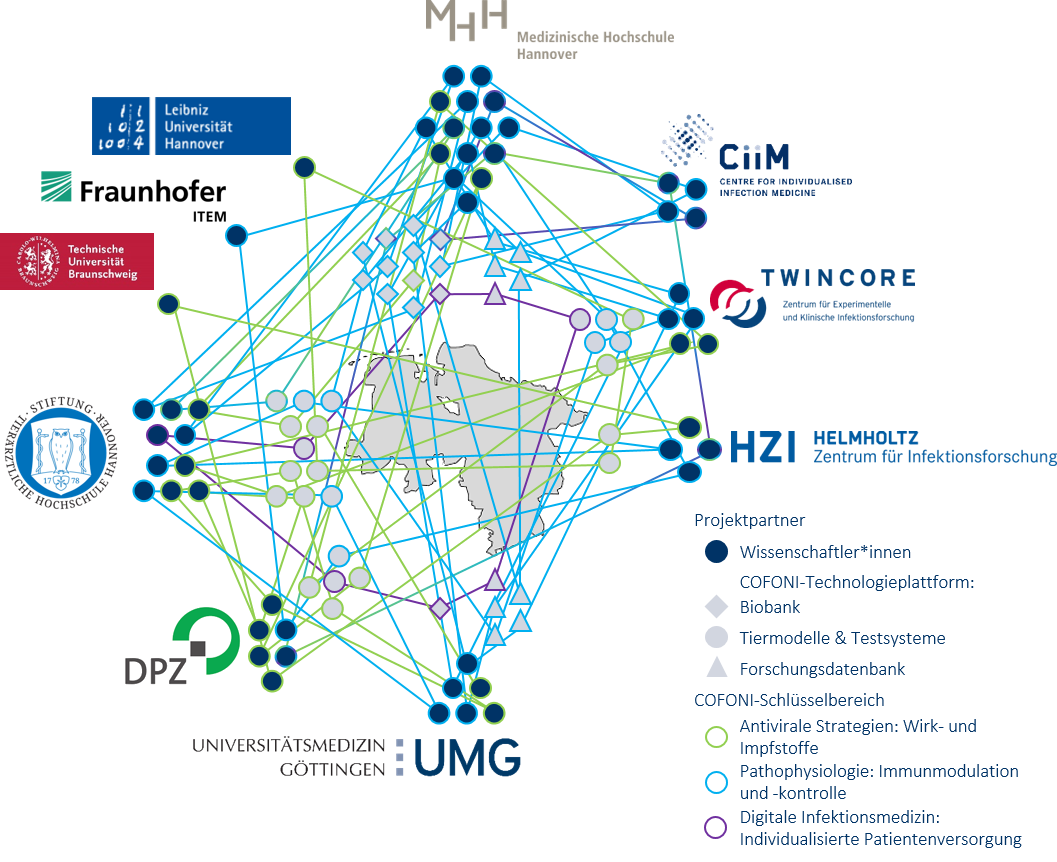
COFONI ist ein Forschungsnetzwerk, das aus fünf niedersächsischen Partnern besteht:
- Georg-August Universität Göttingen und Universitätsmedizin Göttingen,
- Helmholtz Zentrum für Infektionsforschung,
- Medizinische Hochschule Hannover,
- Stiftung Tierärztliche Hochschule Hannover.
- Deutschen Primatenzentrum – Leibniz-Institut für Primatenforschung
Diese Partner möchten ihre wissenschaftlichen Kernkompetenzen zusammenführen, um fünf Schlüsselbereiche zu erforschen, die für die Bewältigung der SARS-CoV-2-Pandemie und ihrer Langzeitfolgen von entscheidender Relevanz sind.
COFONI auf einen Blick
Schlüsselbereiche
Unsere Ziele
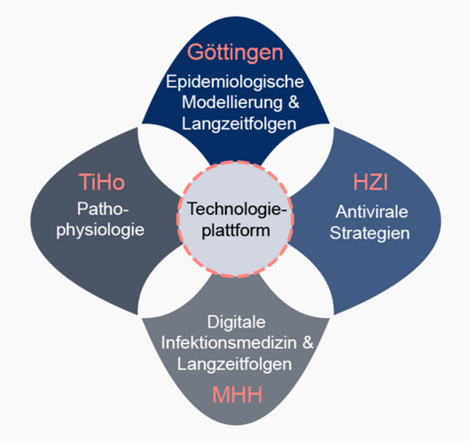
Ziel des Verbundforschungsvorhabens COFONI ist eine koordinierte Zusammenarbeit von Grundlagen-, klinischer und Versorgungsforschung sowie der Sozial- und Gesellschaftswissenschaften, um
- grundlegende und wichtige Fragen zum Virus,
- zu molekularen Grundlagen für die Wirk- und Impfstoffentwicklung,
- zur Vorhersage und Beeinflussung des Pandemiegeschehens sowie
- zur Bewältigung der Langzeitfolgen auf medizinischer, gesellschaftlich-sozialer und psychischer Ebene zu erforschen.
Die neuen Erkenntnisse sollen helfen, neue Therapieformen zu entwickeln und dem Land Niedersachsen weitere Instrumente an die Hand zu geben, um die Auswirkungen der SARS-CoV-2-Pandemie zu bewältigen.
Schlüsselbereich 5
Fördergeber


Projektnetzwerk
So sichern wir Qualität
Zur Sicherung der Qualität der in COFONI durchgeführten Projekte wurden verschiedene Gremien eingerichtet.
Leitungsgremium
Das Leitungsgremium leitet den Forschungsverbund COFONI und stellt eine bedarfsorientiere und wissenschaftlich fundierte Prioritäten- und Schwerpunktsetzung sowie effiziente Mittelverwendung sicher. So stellt es die wissenschaftliche Qualität der geförderten Projektanträge durch ein kompetitives Begutachtungsverfahren sicher und beurteilt die Zielerreichung der Forschungsprojekte. Das Gremium setzt sich wie folgt aus je zwei Beteiligten der Gründungsinstitutionen zusammen:
Prof. Dr. Jürgen Wienands
COFONI-Sprecher
Universitätsmedizin Göttingen

Prof. Dr. Maren von Köckritz-Blickwede
Stellvertretende COFONI-Sprecherin
Stiftung Tierärztliche Hochschule Hannover

Prof. Dr. Mark Brönstrup
Helmholtz-Zentrum für Infektionsforschung

Prof. Dr. Tim Friede
Universitätsmedizin Göttingen

Prof. Dr. Thomas Illig
Medizinische Hochschule Hannover

Prof. Dr. Dr. Michael Marschollek
Peter L. Reichertz Institut für Medizinische Informatik
Medizinische Hochschule Hannover

Prof. Dr. Thomas Pietschmann
TWINCORE – Zentrum für Experimentelle und Klinische Infektionsforschung
Helmholtz-Zentrum für Infektionsforschung

Prof. Dr. Stefan Pöhlmann
Deutsches Primatenzentrum GmbH
Leibniz-Institut für Primatenforschung

Prof. Dr. Asisa Volz
Stiftung Tierärztliche Hochschule Hannover

Externer Beirat
Der externe Beirat stellt die übergeordnete Qualität der Projekte sicher und wird zu den Sitzungen des Leitungsgremiums eingeladen, um bei den Entscheidungen über die geförderten Projekte zu Rate gezogen zu werden. Der Beirat setzt sich aus drei renommierten Fachwissenschaftler*innen zusammen, die nicht am COFONI-Netzwerk beteiligt sind:
Prof. Dr. Markus Bassler
Mitglied des MWK-Expertenrats
Prof. Dr. Sandra Ciesek
Leiterin der Virologie des Universitätsklinikums Frankfurt
Prof. Dr. Dominique Schols
Leiter des Labors für Virologie und Chemotherapie an der KU Leuven, Belgien
Long/Post COVID-Komitee
Eigens zur Qualitätssicherung der geförderten Projekte im Schlüsselbereich „Interdisziplinäre Erforschung der Langzeitfolgen der SARS-CoV-2-Pandemie“ wurde das Long/Post COVID-Komitee gegründet.
Durch die ausgewiesene Fachexpertise der Komitee-Mitglieder in den Bereichen Long/Post COVID, Versorgungsforschung sowie Sozial- und Gesellschaftswissenschaften, berät das Komitee das Leitungsgremium zur Gewährleistung der wissenschaftlich fundierten und versorgungsrelevanten Prioritäten- und Schwerpunktsetzung.
Die personelle Besetzung des Long/Post COVID-Komitees spiegelt ein ausgewogenes Verhältnis der an COFONI beteiligten Wissenschaftsinstitutionen wider sowie eine Schnittmenge zwischen COFONI und dem Long COVID-Expertenrat des Niedersächsischen Ministeriums für Wissenschaft und Kultur.
Kontakt
COFONI-Sprecher*in

Kontaktinformationen
- Telefon: +49 551 3965821
- E-Mail-Adresse: jwienan(at)uni-goettingen.de
Sekretariat
- Telefon: +49 551 3965812
- E-Mail-Adresse: anika.schindler(at)med.uni-goettingen.de

Kontaktinformationen
- Telefon: +49 511 9538787
- Telefax: +49 511 9538585
- E-Mail-Adresse: maren.von.koeckritz-blickwede(at)tiho-hannover.de
COFONI-Koordinierungsstelle

Kontaktinformationen
- Telefon: +49 551 3965878
- E-Mail-Adresse: cofoni(at)med.uni-goettingen.de
Kontaktinformationen
- Telefon: +49 551 3969021
- E-Mail-Adresse: cofoni-lpc(at)med.uni-goettingen.de

Kontaktinformationen
- Telefon: +49 551 3965818
- E-Mail-Adresse: cofoni-finanzen(at)med.uni-goettingen.de
Organisation und Partner
Zentral koordiniert wird die standortübergreifende Vernetzung im Forschungsnetzwerk COFONI durch die Universitätsmedizin Göttingen (UMG). Prof. Dr. Jürgen Wienands, Forschungsdekan der UMG, leitet die zentrale Koordinierungsstelle zusammen mit Prof. Dr. Maren von Köckritz-Blickwede von der Stiftung Tierärztliche Hochschule Hannover (TiHo).
Downloads