Neue bildgebende Methode ermöglicht Identifizierung seltener Synapsen im Gehirn

Die Komplexität lebender Systeme ist das Ergebnis komplizierter Netzwerke, in denen verschiedene biomolekulare Prozesse genau aufeinander abgestimmt sind, um Zellen, Gewebe und ganze Organismen zu bilden. Um ein umfassendes Verständnis dieser biologischen Komplexität zu erlangen, müssen die einzelnen Biomoleküle identifiziert, ihr Vorkommen im Körper und ihre Wechselwirkungen miteinander bestimmt werden. Die Erfassung dieser Komplexität mit Nanometerauflösung für mehrere Moleküle gleichzeitig ist eine aktuelle wissenschaftliche und technologische Herausforderung. Die überwiegende Mehrheit der zellulären Funktionen wird von Proteinen ausgeführt. Proteine sind Biomoleküle, die für die Form und den Aufbau der Zellen sowie für die biochemischen Stoffwechselprozesse zuständig sind. Bisher gab es keine Technologie, mit der man mehr als 15 Proteinspezies bei einer sehr hohen Auflösung leicht erkennen konnte.
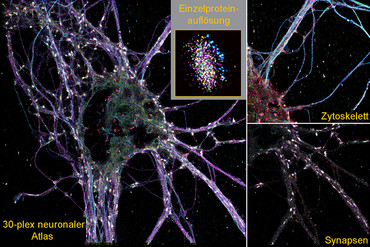
Wissenschaftler*innen um Dr. Eugenio F. Fornasiero, Arbeitsgruppenleiter am Institut für Neuro- und Sinnesphysiologie der Universitätsmedizin Göttingen (UMG), Dr. Felipe Opazo, Arbeitsgruppenleiter am Institut für Neuro- und Sinnesphysiologie und am Center for Biostructural Imaging of Neurodegeneration (BIN) der UMG, Prof. Dr. Ralf Jungmann, Leiter der Forschungsgruppe „Molekulare Bildgebung und Bionanotechnologie“ am Max-Planck-Institut für Biochemie (MPIB) in Martinsried und Inhaber des Lehrstuhls für Molekulare Physik des Lebens an der Ludwig-Maximilians-Universität München (LMU), und Dr. Carsten Marr, Direktor des Institute of AI for Health am Helmholtz Munich, haben gemeinsam ein neues hochauflösendes bildgebendes Verfahren entwickelt, mit demdie Vielfalt der Gehirnsynapsen untersucht werden kann. Synapsen sind die Kontaktstellen zwischen den Nervenzellen und dienen der Informationsweiterleitung. Mit ihrer neu entwickelten Technologie, der sogenannten SUM-PAINT-Methode, konnten die Forschenden 30 verschiedene Proteine in den Nervenzellen des Gehirns hochauflösend in dreidimensionaler Struktur (3D) gleichzeitig darstellen und ihr Zusammenspiel analysieren. Durch diese Form der gleichzeitigen Beobachtung von Molekülen, auch Multiplexing genannt, konnte die Komplexität der synaptischen Proteinzusammensetzung von fast 900 Synapsen entschlüsselt und eine neue Art seltener Synapsen im Gehirn identifiziert werden. Die Ergebnisse sind in der renommierten Fachzeitschrift Cell publiziert.
Originalpublikation:
Spatial Proteomics at single-protein resolution. Eduard M. Unterauer*, Sayedali Shetab Boushehri*, Kristina Jevdokimenko*, Luciano A. Masullo, Mahipal Ganji, Shama Sograte-Idrissi, Rafal Kowalewski, Sebastian Strauss, Susanne C.M. Reinhardt, Ana Periodic, Carsten Marr, Felipe Opazo, Eugenio F. Fornasiero§, Ralf Jungmann§. 2024 Cell 187, 1-16, March 28. DOI: https://doi.org/10.1016/j.cell.2024.02.045
* = shared first authors, § = correspondence
„Die SUM-PAINT-Methode kann relativ einfach mit handelsüblicher Mikroskop-Hardware kombiniert werden und liefert einen detaillierten Überblick über die Lokalisation und Interaktion einer großen Anzahl von Proteinen auf molekularer Ebene. Dies ermöglicht es, bisher schwer erfassbare Details neurologischer Störungen zu untersuchen und könnte zu einem tieferen Verständnis der zugrunde liegenden Krankheitsmechanismen beitragen“, sagt Dr. Eugenio F. Fornasiero von der UMG und einer der Letztautoren der Studie. „Ich bin davon überzeugt, dass diese Technologie entscheidend dazu beitragen wird, bisher unbekannte Veränderungen im Nervensystem zu erforschen, die zu neurodegenerativen Erkrankungen führen“, so Dr. Fornasiero.
"Dies ist ein Technologiesprung in der superauflösenden Mikroskopie. Wir können die räumliche Anordnung sowie Interaktion von 30 Proteinen oder auch anderer Zellbestandteile gleichzeitig in einem Bild erfassen. Durch die Kombination mehrerer Informationsarten in einem einzigen Bild kann ein umfassenderes Bild einer Probe gewonnen werden, ohne für jedes Merkmal separate Bilder aufnehmen zu müssen. Dies ermöglicht eine effizientere Analyse von biologischen Proben bis zu hundertmal schneller als mit bisherigen Mikroskopietechniken", sagt Eduard Unterauer, Erstautor der Studie aus Prof. Jungmanns Gruppe.
Details zur Studie
Die Untersuchung von Proteinzusammensetzungen wurde durch superauflösende Mikroskopietechniken erheblich vorangebracht. Viele dieser Techniken können jedoch nicht mehr als eine Handvoll Zielmoleküle in hoher Auflösung abbilden. Die secondary label-based unlimited multiplexed (SUM)-PAINT-Methode basiert auf der DNA-PAINT-Methode, die von der Forschungsgruppe um Prof. Dr. Ralf Jungmann entwickelt wurde. Hierbei handelt es sich um eine Superauflösungstechnik, die Strukturen kleiner als 20 Nanometer sichtbar macht. Dies ist etwa 2.500 Mal kleiner als der Durchmesser eines menschlichen Haares. Es werden kurze farbstoffmarkierte DNA-Stränge verwendet, die vorübergehend an ihre Zielmoleküle binden und ein "Blinken", wie ein kleines Funkeln in der Dunkelheit, erzeugen. Durch dieses Blinken können die Zielmoleküle in der Zelle dargestellt und lokalisiert werden. Diese Informationen werden zusammengefügt und ergeben so die superauflösenden Bilder.
Um die Möglichkeiten von SUM-PAINT zu testen und auf einen zellulären Kontext anzuwenden, haben die Gruppen von Dr. Fornasiero und Dr. Opazo an der UMG in enger Zusammenarbeit die Strategien für eine zuverlässige Markierung von Proteinen in Nervenzellen getestet, optimiert und verfeinert. Zur Darstellung der Proteine in den Nervenzellen war der Einsatz von Antikörpern und den noch viel kleineren Nanokörpern von besonderer Bedeutung. Diese waren mit kurzen DNA-Codes markiert und ermöglichten es, gezielt bestimmte Proteine in den Nervenzellen zu erkennen und zu lokalisieren. Analog zur DNA-PAINT-Methode werden die Lage und Struktur der Zielproteine durch „Blinken“ sichtbar gemacht. Diese Optimierungsarbeiten waren der Schlüssel zum ersten neuronalen Atlas, der 30 verschiedene Proteine innerhalb der Nervenzellen charakterisiert. Dies wurde ermöglicht durch die Unterstützung der Göttinger Unternehmen NanoTag Biotechnologies und Synaptic Systems.
Insgesamt konnte die Komplexität der synaptischen Proteinzusammensetzung von fast 900 Synapsen entschlüsselt werden. Um die inhaltsreichen Datensätze zu untersuchen, wurden verschiedene Analysewerkzeuge basierend auf maschinellem Lernen entwickelt und systematisch zur Datenanalyse eingesetzt. Beim maschinellen Lernen wird die künstliche Intelligenz darauf trainiert, Datensätze zu analysieren, um versteckte Regeln abzuleiten, die mit dem menschlichen Auge nicht zu erkennen wären. Durch die Extraktion von 1.600 Merkmalen aus den Bilddatensätzen wie beispielsweise Proteingehalt, -verteilung und -form konnten die Forschenden einen neuen, bisher unbekannten Typ von Synapsen entdecken.
Kristina Jevdokimenko, Doktorandin im Fornasiero-Labor der UMG und ebenfalls Erstautorin der Studie, konnte im Hirngewebe zeigen, dass diese Synapsen aufgrund ihrer einzigartigen Proteinzusammensetzung nur etwa ein Prozent aller Synapsen im Gehirn ausmachen und mit anderen bildgebenden Verfahren nicht entdeckt worden wären. Das Team hat nicht nur einen neuen Typ von Synapsen entdeckt und einen bisher unerreichten biologischen Einblick in die synaptische Vielfalt ermöglicht, sondern auch einen integrierten Arbeitsablauf für die hochkomplexe Datengenerierung und -analyse bereitgestellt. Dieser steht Forschenden auf der ganzen Welt zur Verfügung, um ein bisher unerreichtes Verständnis ihrer spezifischen biologischen Forschungsfragen zu erlangen.
KONTAKT
Universitätsmedizin Göttingen, Georg-August-Universität
Institut für Neuro- und Sinnesphysiologie
Dr. Eugenio F. Fornasiero
Humboldtallee 23, 37073 Göttingen
Telefon 0551 / 39-67930
eugenio.fornasiero(at)med.uni-goettingen.de
fornasierolab.uni-goettingen.de
Universitätsmedizin Göttingen, Georg-August-Universität
Center for Biostructural Imaging of Neurodegeneration (BIN)
Dr. Felipe Opazo
Von-Siebold-Straße 3a, 37075 Göttingen
Telefon 0551 / 39-61156
felipe.opazo(at)med.uni-goettingen.de
opazolab.de
Universitätsmedizin Göttingen, Georg-August-Universität
Leitung Unternehmenskommunikation
Lena Bösch (Pressekontakt UMG)
Von-Siebold-Straße 3, 37075 Göttingen
Telefon 0551 / 39-61020, Fax 0551 / 39-61023
presse.medizin(at)med.uni-goettingen.de
www.umg.eu